
Mise en évidence de la transcription et/ou de la traduction d’un gène
Sommaire
| 1. Considérations générales | Go ! |
| A. Principe de l'utilisation du gène de la luciférase de luciole | Go ! |
| B. Caractéristiques et avantages de l'utilisation de la luciférase de luciole | Go ! |
| 2. Réalisation pratique : utilisation de vecteurs génétiques | Go ! |
| A. Définition et principe de l'utilisation d'un vecteur | Go ! |
| B. Réalisation de l'insertion du gène | Go ! |
| C. Sélection et utilisation des vecteurs recombinants | Go ! |
| D. Limites de la méthode et problèmes rencontrés | Go ! |
| 3. Applications et expériences : exemples de recherche de l'expression de gènes | Go ! |
| A. Mesure de la transcription chez les animaux | Go ! |
| a. Expérience de l'artère de chien | Go ! |
| b. Mesure de la transcription dans une Drosophile vivante | Go ! |
| B. Etudes chez les végétaux | Go ! |
| a. Le début des expériences sur les plantes | Go ! |
| b. Etude de la régulation des rythmes circadiens | Go ! |
| C. Applications sanitaires | Go ! |
| a. Le test de la tuberculose | Go ! |
| b. Autres applications sanitaires et médicales | Go ! |
A. Principe de l’utilisation du gène de la luciférase de luciole
Le gène de la luciférase (noté luc) est utilisé en biologie moléculaire en tant que gène marqueur. Inséré à l’intérieur d’un autre gène, si ce gène est actif, le gène marqueur luc est transcrit et traduit en même temps que le gène dans lequel il a été inséré. En milieu contenant de la luciférine et de l’ATP, la luciférase ainsi synthétisée est responsable d’une émission lumineuse(si le gène considéré n’est pas actif, il n’y a pas d’émission lumineuse). Comme toutes les enzymes ne sont pas facilement détectables, les gènes marqueurs sont utilisés à la place des gènes codant pour les enzymes. Ces gènes marqueurs amènent à la mesure de l’activité de la protéine correspondante.

Les gènes marqueurs sont utilisés le plus souvent pour analyser et préciser le fonctionnement d’éléments régulateurs du génome (facteurs cis = séquences régulant l’activité d’un gène) comme les activateurs ou les promoteurs, responsables du contrôle de la production d’enzymes. Dans des expériences classiques, des délétions ou des mutations sont pratiquées dans une région promotrice, et leurs effets sur le gène concerné couplé au gène marqueur sont observés et quantifiés.
L’utilisation du gène luc en tant que marqueur est courante à cause de la rapidité de la réaction de bioluminescence, de sa sensibilité et de sa simplicité d’exécution. L’utilisation de marqueurs produisant de la lumière est recommandée à cause de l’absence de déchets, contrairement aux techniques de mesures de radioactivité par exemple. De plus, l’appareil de mesure, le luminomètre, est très sensible et peut mesurer des quantités d’enzyme aussi faibles que 10 ou 20 moles. La quantification de l’expression du gène est donc possible par simple mesure de l’intensité lumineuse. En outre, par cette méthode, on s’affranchit du "bruit de fond " rencontré lors de l’utilisation d’autres méthodes de dosage d’activité enzymatique ou moléculaire : il n’y a aucune activité endogène au sein des cellules utilisées qui pourrait interférer avec la mesure de l’intensité lumineuse.
Ainsi, le rôle des marqueurs génétiques est de permettre une rapide évaluation quantitative d’événements physiologiques sans coût ni effort extraordinaire. La luciférase de luciole est presque idéale pour cette utilisation, ainsi que nous allons l’expliquer par la suite.
B. Caractéristiques et avantages de l’utilisation de la luciférase de luciole
La bioluminescence est présente dans de nombreux phyla, particulièrement en milieu marin : bactéries, annélides, mollusques, dinoflagellés, poissons, mais aussi champignons et insectes. Chacun de ces groupes a évolué de manière différente au niveau biochimique, et en ce qui concerne la production de lumière, des systèmes chimiques très différents se sont mis en place. Néanmoins, le choix de l’utilisation du gène de telle ou telle luciférase en tant que gène marqueur est plutôt réduit car peu ont été étudiées en détails. Seules les luciférases du ver luisant, de Renilla reniformis et des bactéries sont utilisables au niveau pratique pour le moment. Les autres n’ont pas trouvé d’application pratique soit à cause de la difficulté à synthétiser le substrat de la réaction, soit parce que la réaction demande un catalyseur handicapant au niveau expérimental.

La luciférase du ver luisant est la plus communément utilisée. La lumière émise par la réaction d’oxydation qu’elle catalyse est verte ou jaune (550-570 nm). Plusieurs stratégies ont été mises au point afin de stabiliser le signal lumineux, car celui-ci décroît assez vite après une phase très intense qui dure environ 15 secondes. Par exemple, l’ajout de coenzyme A permet d’obtenir un signal qui dure quelques minutes, et dont l’intensité permet au luminomètre d’obtenir une sensibilité inférieure à 10 moles d’enzymes.
Ce qui a déterminé le choix de la luciférase de luciole est le fait que le gène luc codant pour cette luciférase soit un ADNc (ADN complémentaire d’un ARNm mature), et que par conséquent il ne contient pas d’intron. On peut donc l’utiliser à la fois chez les eucaryotes et les procaryotes, puisque l’ARN transcrit à partir de cet ADN ne nécessite aucune maturation. La luciférase est un monomère qui ne requiert aucune modification post-traductionnelle, et elle est donc disponible en tant qu’enzyme fonctionnelle directement après la traduction de son ARNm. On a d’ailleurs montré que son action catalytique est possible dès la sortie du ribosome. Ainsi, les expériences avec la luciférase offrent des résultats instantanés qui traduisent la totalité de l’expression des gènes actifs dans la cellule.
L’utilisation simultanée de luciférase de luciole et de Renilla reniformis peut permettre de séparer deux événements transcriptionnels au sein du même système, étant donné que les deux enzymes possèdent des promoteurs différents. L’adjonction d’un inhibiteur peut être révélée grâce à la non-transcription de l’une ou l’autre des deux luciférases dont le mécanisme biochimique enzymatique n’est pas le même et qui n’émettent donc pas exactement à la même longueur d’onde. Les deux luciférases sont complémentaires, mais en définitive, la luciférase de Renilla reniformis n’est en général pas recommandée en tant qu’alternative à la luciférase de luciole, même si elle en possède certaines qualités. La luciférase de Renilla reniformis peut servir de compagnon à la luciférase de luciole, dans les seuls cas où deux gènes marqueurs sont nécessaires. Les marqueurs sont souvent utilisés en tandem afin d’augmenter la précision de l’analyse de l’activité du gène luc. L’avantage d’une telle méthode est de posséder un contrôle (l’activité de la luciférase de Renilla) qui normalisera l’activité de la luciférase de luciole. Les sources de variabilité comme les différences dans les techniques manuelles, le pipettage et les lyses cellulaires peuvent être éliminées. La réponse normalisée est calculée comme suit :
| marqueur " expérimental " (luciole) | |
| Luminescence normalisée = | ---------------------------------- |
| marqueur " de contrôle " (Renilla) | |
En théorie, des protéines non luminescentes peuvent aussi être utilisées en tant que premier ou deuxième marqueur, mais les techniques de détection du deuxième gène marqueur, comme la colorimétrie, la radiométrie ou la fluorimétrie, entraînent une perte de temps conséquente par rapport à la luminométrie. C’est pourquoi il est pratique d’utiliser deux marqueurs bioluminescents.
2. Réalisation pratique : utilisation de vecteurs génétiques
A. Définition et principe de l’utilisation d’un vecteur
Un vecteur est une séquence d’ADN de petite taille, capable de s’auto-répliquer, d’être transcrite et traduite, et dans laquelle on peut insérer le gène qui nous intéresse (ici le gène luc). On en distingue plusieurs types : les plasmides bactériens, les virus (surtout les bactériophages) et les cosmides.
Le but de l’utilisation d’un vecteur est d’obtenir une amplification importante par copie du gène inséré. C’est une méthode pratique pour fabriquer des quantités suffisantes de matériel avec lequel il est possible de travailler.
Un vecteur doit posséder un polylinker, c’est à dire une zone de l’ADN où se trouvent des sites uniques de restriction, ainsi qu’un gène de sélection qui permettra de reconnaître les vecteurs recombinant (qui auront intégré le gène d’intérêt, ici le gène luc) des vecteurs non recombinant.
Le vecteur doit aussi contenir le gène dont on désire connaître l’activité (on le nommera par la suite " act ? ") et qui va être couplé au gène luc. En pratique, on va insérer le gène act ? en premier, et on insérera le gène luc au milieu du gène act ?. De cette manière, si le gène act est actif, comme celui-ci a été inséré avec son promoteur, la transcription va avoir lieu jusqu’au site de terminaison, et l’ARN qui en résultera aura cette structure :
(5") act-luc-act (3")
Ainsi, si le gène luc est traduit, la luciférase sera active car l’exon est entier.
Il est également possible que le vecteur contienne des sites régulateurs qui ont été insérés , et qui vont agir directement sur la transcription du gène luc.
B. Réalisation de l’insertion du gène
Nous prendrons l’exemple d’un plasmide, car c’est le vecteur le plus utilisé.
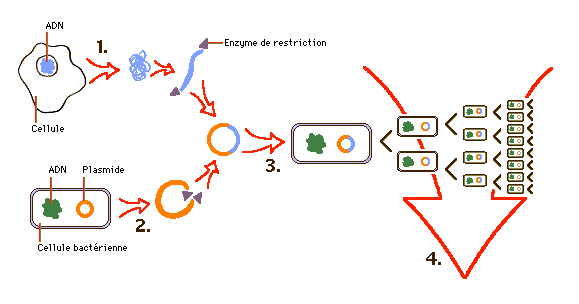
(Microsoft Encarta)
L’ADN du gène luc à insérer est extrait de la cellule de ver luisant, et isolé à l’aide d’enzymes de restriction reconnaissant des séquences particulières situées au début et à la fin du gène (étape 1 sur la figure).
Le plasmide est lui aussi extrait de sa bactérie, et on le coupe en un seul point à l’aide d’une enzyme de restriction (d’où l’intérêt des sites uniques de restriction du polylinker). Grâce à des ligases, on insère le gène luc dans le plasmide (étape 2).
Les bactéries vont alors intégrer le vecteur recombinant par transformation bactérienne (étape 3) une fois qu’elles auront été rendues compétentes par un traitement chimique qui altère la structure de la paroi bactérienne ou par électroporation. Cette transformation se fait le plus souvent à l’aide d’un choc thermique (30 mn à 0°C puis 45 s à 42 °C). Le plasmide traverse alors la membrane plasmique et va être répliqué plusieurs fois à l’intérieur de la bactérie sans que celle-ci se divise, ainsi que lors des divisions binaires bactériennes (étape 4), après incubation à une température variable suivant le type de bactérie utilisé. Voici un exemple de plasmide obtenu après insertion du gène luc de Renilla reniformis :

C. Sélection et utilisation des vecteurs recombinant
La première étape de l’utilisation des plasmides est la détermination des bactéries transformant (qui ont intégré un plasmide non recombinant) et des bactéries transformant recombinant (qui ont intégré des plasmides recombinant, i.e. contenant le gène luc), par rapport aux bactéries non transformant. Pour cela on utilise un gène de sélection. Le plasmide ci-dessus possède un gène " Ampr ", qui procure à la bactérie un caractère de résistance à un antibiotique : l’ampicilline. En milieu contenant de l’ampicilline, seules les bactéries ayant intégré le plasmide pourront survivre. Ensuite pour différencier les transformant des non transformant, on utilise un autre gène de sélection, par exemple le gène LacZ (non présent ici) qui a la propriété de produire une substance bleue s’il est transcrit et traduit. Le gène luc étant inséré au milieu du gène LacZ, celui-ci ne peut être fonctionnel que dans le cas où le plasmide ne serait pas recombinant. Au final, dans un milieu contenant de l’ampicilline et le substrat pour le gène LacZ (X-gal et IPTG), on sélectionnera les colonies bactériennes blanches qui seront recombinant (les bleues ne seront que transformant).
Une fois les bactéries sélectionnées, il va falloir purifier les vecteurs, afin que l’ADN soit exempt de toute protéine, de tout ARN ou d’une quelconque contamination chimique. Puis, avant de commencer les expériences proprement dites, on transfecte les plasmides dans les cellules qui nous intéressent.
La transfection est le processus par lequel des acides nucléiques exogènes sont introduits dans la cellule en culture dont on désire étudier une voie métabolique, que ce soit, dans des cellules de Mammifères, par des moyens physiques :
| - micro-injection cellulaire de matériel, | |
| - électroporation qui entraîne la formation de pores dans la membrane cellulaire et qui permet ainsi à l’ADN de pénétrer dans le cytoplasme ; | |
ou biochimiques :
| - utilisation de phosphate de calcium qui précipite avec l’ADN ; ce précipité adhère à la membrane plasmique et est endocyté par la membrane cellulaire, | |
| - utilisation de lipides cationiques formant des liposomes chargés + attirant à la fois l’ADN chargé – et la membrane plasmique. |
Une fois le plasmide intégré dans le cytoplasme des cellules en culture, on va pouvoir détecter et suivre l’expression du gène luc en milieu approprié (luciférine et ATP notamment), et ainsi déterminer les portions du gène couplé au gène luc qui sont responsables de régulations transcriptionnelles. La mesure des émissions lumineuses se fait grâce au luminomètre que nous avons vu précédemment.
D. Limites de la méthode et problèmes rencontrés
Il existe plusieurs inconvénients à cette méthode, dus pour la plupart aux incertitudes des manipulations expérimentales touchant les plasmides. Le taux de recombinant n’est pas de 100% au niveau de l’insertion de l’ADN des gènes à étudier, et de plus l’utilisation de plusieurs enzymes de restriction fait augmenter la part du hasard dans la réussite de la manipulation. C’est pourquoi l’étape d’amplification est essentielle pour maximiser la quantité de matériel utilisable au final.
D’autre part, l’efficacité de la transfection est liée de très près à la qualité des réactifs utilisés, et en cas de faible activité de la luciférase dans tous les échantillons, il va être nécessaire de reconsidérer les conditions d’expérimentation ou de changer de type de plasmide. On peut également vérifier que les cultures ne sont pas contaminées avec des micro-organismes, et notamment des Mycoplasmes.
Il est également possible que l’on obtienne des faux négatifs, c’est à dire le niveau d’activité de la luciférase est élevé au niveau des témoins de contrôle, mais faible lors des essais après cotransfection avec le gène d’intérêt act ?.Il s’agit alors d’un problème de pureté au niveau de l’ADN du plasmide. La solution est de changer de plasmide ou de méthode de transfection, voire les deux.
3. Applications et expériences : exemples de recherche de l’expression de gènes
A cause de la facilité de détection de la lumière par un luminomètre, les applications de la bioluminescence dans le domaine de la recherche scientifique sont en train de se multiplier rapidement. Celles qui suivent n’en représentent qu’un petit nombre, parmi lesquelles certaines n’ont été développées que très récemment.
A. Mesure de la transcription chez les animaux
a. Expérience de l’artère de chien
En 1991, la bioluminescence a été pour la première fois induite artificiellement dans des cellules de Mammifère. Les artères d’un chien ont été mises en contact avec des plasmides bactériens contenant des gènes codant pour la luciférase, afin de déterminer si des gènes étrangers pouvaient être incorporés dans le génome du chien. Le choix du gène codant pour la luciférase était ingénieux à cause de sa facilité de détection. Après avoir laissé aux artères le temps nécessaire à l’incorporation du gène luc, les scientifiques menant l’expérience ont simplement ajouté de la luciférine pour voir si la luciférase était effectivement synthétisée. L’émission lumineuse qui accompagne l’addition de luciférine a bien semblé le prouver. Les résultats ont été encourageants car une fois que la possibilité d’insérer des gènes dans le génome des cellules de Mammifères a été prouvée, les applications médicales ont pu être envisagées, et notamment l’insertion de gènes produisant des molécules pouvant dissoudre les caillots dans des artères humaines de patients à haut risque d’infarctus.
b. Mesure de la transcription dans une Drosophile vivante
La luciférase en tant que gène marqueur combiné avec un système d’imagerie vidéo à faible luminosité offre un moyen d’étudier des fluctuations dynamiques dans une quelconque activité transcriptionnelle. La faible intensité de la lumière produite peut être mesurée par imagerie vidéo à condition d’utiliser les lentilles appropriées ; sous microscope, il est possible d’atteindre une résolution de l’ordre de la cellule.
Plusieurs applications évidentes combinant l’expression de la luciférase et les technologies de l’imagerie concernent un grand éventail d’organismes. Des chercheurs se sont plus particulièrement intéressés à l’étude de la régulation circadienne de la transcription chez la Drosophile, chez qui l’horloge circadienne a été sujette à une analyse génétique détaillée. Finalement, le but de l’utilisation de la luciférase chez cette mouche est de développer les outils nécessaires pour mesurer la transcription de gènes neuronaux et surveiller en même temps le comportement de l’animal, et de ce fait obtenir des corrélations moléculaires au niveau du contrôle de la fonction complexe du cerveau.
Les mouches utilisées sont des mouches transgéniques exprimant le gène luc de manière intense. Pendant ces expériences, on a découvert deux moyens efficaces pour faire incorporer la luciférine à l’organisme : une simple pulvérisation ou l’inclusion de la luciférine dans la nourriture de l’animal.

La photo montre qu’il est facile de mesurer la luminescence dans un animal vivant et anesthésié. Mais plusieurs facteurs peuvent affecter les résultats de ces mesures chez la Drosophile : la pigmentation du corps peut occulter la lumière verte émise dans certaines parties du corps, comme c’est le cas ici au niveau des yeux. Lorsque l’on utilise des mutants portant la mutation " yeux blancs ", on observe que la région des yeux est presque la plus lumineuse de l’organisme. C’est donc ce type de mouche qu’il faut utiliser pour ne pas voir les résultats faussés par un faux négatif, ou des mouches portant toute autre mutation réduisant la pigmentation.
Enfin, il faut noter qu’aucun effet délétère dû aux expositions prolongées et répétées à la luciférine n’a été constaté sur les animaux.
En résumé, on peut dire que l’utilisation de la luciférase combinée à des systèmes d’imagerie à faible luminosité possède un potentiel unique pour étudier à la fois la régulation temporelle et spatiale de la régulation de l’expression des gènes chez des organismes pluricellulaires vivants. Cette technologie pourrait bientôt être applicable à tout système génique transparent à la lumière verte et accessible à la luciférine exogène. De plus l’étude peut être étendue à de plus larges populations qu’un organisme isolé, ce qui ouvre la voie à de nombreuses possibilités d’isolement de mutants présentant des phénotypes de luminescence aberrante par exemple.
a. Le début des expériences sur les plantes
Le royaume des plantes a aussi subi des expériences de bioluminescence induite par la recherche scientifique. Il y a quelques années, les généticiens ont entrepris de transplanter de l'ADN dans les plantes. La paroi cellulaire des cellules végétales a posé de gros problèmes et a rendu l'opération de transplantation très délicate. De plus, dans les quelques plantes transplantées avec succès, l'ADN fut le plus souvent détruit ou rejeté par les cellules. Et même si une cellule eut accepté l'ADN, celui-ci restait ponctuel dans l'organisme, et les chercheurs voulaient obtenir une plante entièrement transgénique. D'où la nécessité d'utiliser des vecteurs de gènes. Après plusieurs années d'essais infructueux, les généticiens étudièrent la possibilité d'infecter les plantes avec la bactérie Agrobacterium tumefaciens présente dans le sol. Cette bactérie, en infectant une cellule végétale, insère un morceau d'ADN utilisé par la cellule, de la même manière que les virus manipulent les cellules pour se reproduire. Cet ADN stimule de même la croissance et la division de la cellule, éventuellement jusqu'à la formation d'un adulte si les conditions requises sont respectées. La seule différence est qu'après le départ de la bactérie, la plante utilise toujours cet ADN.
Afin de tester cette nouvelle technique de formation de plantes transgéniques, les scientifiques ont utilisé une protéine luminescente facile à tester et inoffensive pour la plante. Les laboratoires ont utilisé la bioluminescence car par ce moyen, il est évident de voir si l'expérience est réussie ou non. Au début, le succès était partiel car le gène n'était pas idéal pour l'expérience. En 1990, la bactérie Agrobacterium tumefaciens fut utilisée pour la première fois pour introduire le gène luc dans des cellules de plants de tabac. Le résultat fut un plant de tabac qui brillait d'une lumière verte après administration du substrat, la luciférine.
b. Etude de la régulation des rythmes circadiens
Comme chez la Drosophile, on a également utilisé la bioluminescence pour étudier les gènes responsables du rythme circadien des plantes. Chez les animaux, les gènes gouvernant ces cycles ont été localisés plus facilement car les scientifiques ont pu comparer les génomes d'animaux possédant une horloge biologique normale avec ceux d'animaux ayant des horloges anormales ou mutantes. En comparant les deux génomes, on peut trouver ces gènes circadiens en cherchant des différences inexpliquées. Mais les scientifiques ont eu beaucoup plus de mal à trouver de plantes possédant des horloges mutantes qu'à trouver des hamsters restant éveillés toute la nuit et dormant le jour. Pour mieux localiser ces plantes mutantes, le gène codant pour la luciférase a été inséré dans le génome de plusieurs plants, et le gène a été couplé à une séquence régulatrice que l'on savait activée le jour et inactivée la nuit par le gène de l'horloge circadienne. Lors de l'ajout de luciférine, les mutants ont étés facilement identifiables par le fait qu'ils produisaient de la lumière la nuit, au lieu du jour comme les autres. Ceci indique que leur horloge circadienne mutante envoie un mauvais signal à la séquence régulatrice à laquelle le gène luc inséré a été couplé.
D'autres études ont été entreprises, notamment autour du gène cab2, qui appartient à une famille de gènes codant pour des protéines intervenant dans les complexes photosynthétiques. De nombreuses voies de signalisation cellulaire interviennent dans la régulation des gènes cab, et dans cet exemple on s'est intéressé à la régulation du gène cab2 par la lumière et les rythmes de l'horloge circadienne. A cause de la nécessité de mesurer l'expression des gènes de manière extrêmement fréquente, il a fallu trouver des méthodes plus fiables que l'utilisation des ARNm (on dose les ARNm à intervalles réguliers pour déterminer l'ampleur de la transcription du gène d'intérêt). En plus de ne pas présenter ces inconvénients, l'utilisation de la luciférase offre l'avantage d'avoir une activité relativement instable in vivo, ce qui permet d'en étudier facilement les variations avec une précision satisfaisante, et de manière indépendante a l'aide de l'imagerie à comptage de photon (luminomètre ou caméra vidéo à faible luminosité).
Les essais ont été entrepris avec Arabidopsis thaliana. Environ 500 plants transgéniques (on a inséré le gène luc à côté du gène cab2) ont été cultivés après germination pendant 7 jours, avec des cycles alternant 12 heures de lumière et 12 heures d'obscurité. Les plants furent arrosés avec de la luciférine trois fois à 6 heures d'intervalle avant d'être filmés. A chaque fois, la luminescence fut enregistrée pendant un laps de temps de 25 minutes.
La photo qui suit montre la luminescence émise à 11h00 et 23h00 dans un cycle lumière / obscurité pour les photos du haut et à la seule lumière pour les photos du bas. Elle prouve que le gène cab2 est surtout actif le jour.

Une image identique obtenue avec des plants de tabac transgéniques a montré que le fragment contenant le gène cab2 confère un rythme circadien robuste dont la luminescence fut facilement enregistrée pendant plusieurs jours.

Une analyse équivalente basée sur l'extraction et la protection d'ARN aurait requis une bien plus grande quantité de tissus végétaux et aurait été beaucoup plus laborieuse. On a également tiré avantage de la facilité d'utilisation de la luciférase pour conduire une analyse détaillée des éléments régulateurs présents dans le promoteur du gène cab2 responsables de la régulation circadienne de la transcription. De plus, l'activité de la luciférase a permis d'identifier les mutants dans les horloges circadiennes des plants d'Arabidopsis.
Les images enregistrées sur de jeunes plants ont également montré que la luminescence était émise spécifiquement dans les cotylédons, et ce résultat reflète une spécificité tissulaire d'expression du gène cab. En effet, en plus de permettre l'enregistrement de la régulation du gène au cours du temps, la luciférase permet aussi d'étudier la régulation spatiale du gène cab. Sur la photo qui suit, on voit que la luminescence des feuilles est majoritairement émise au niveau des nervures.

Il a été possible de montrer que la luminescence observée dans les feuilles évoluait de manière cyclique, alors que la luminescence dans les racines n'oscillait pas. Ceci suggère que l'horloge du gène cab2 et les éléments régulateurs des systèmes photosynthétiques impliqués dans le rythme circadien ne fonctionnent pas dans les racines. La possibilité de mesurer les composants spatio-temporels de la transcription dans des tissus vivants offre un outil puissant pour l'étude des événements moléculaires sous-jacents qui régulent le développement.
Poser un diagnostic de tuberculose a longtemps posé un problème à cause de la procédure de détermination des souches : en effet, la méthode courante consiste à cultiver de manière importante la souche de Mycobacterium tuberculosis particulière du patient, opération qui peut prendre 3 à 8 semaines. La culture est alors testée avec les 11 médicaments disponibles, afin de voir lequel est efficace. Cette opération peut prendre 5 semaines. Bien sûr, ces délais posent un problème, car souvent le patient ne peut attendre le diagnostic, et si on lui administre l'un des 11 médicaments, la probabilité est faible de lui prescrire le bon sans avoir déterminé la souche de la bactérie.
Une récente alternative à ces délais s'est mise en place et consiste à incorporer la bioluminescence dans le test de la tuberculose (TB). Cette méthode, qui sera bientôt parfaitement au point, va réduire de manière conséquente le temps nécessaire au diagnostic et révéler la résistance de la souche contractée par le patient aux anti-tuberculineux dans un délai de deux jours. La technique consiste à insérer le gène de luciole qui code pour la luciférase dans le génome de la culture de bactéries TB prélevées sur le patient. Le gène luc est introduit par un vecteur viral et une fois incorporé, la bactérie produit la luciférase. Lors de l'ajout de luciférine dans le milieu de culture, de la lumière est émise. Comme moins de 10 000 bactéries sont nécessaires pour produire suffisamment de luciférase émettant une lumière détectable par un luminomètre, le temps de culture est réduit à seulement 2 ou 3 jours. La réaction luciférine-luciférase nécessitant de l'ATP, la résistance de la souche de culture peut être testée en ajoutant le médicament et en analysant la lumière émise. Si de la lumière est détectée par le luminomètre, la souche est résistante car elle est toujours vivante et produit de l'ATP. Si aucune n'est émise, la bactérie doit avoir été tuée par le médicament et ne produit plus l'ATP nécessaire à la réaction de bioluminescence. En réduisant le temps nécessaire à la prescription du traitement approprié, cette application de la bioluminescence va bientôt pouvoir sauver une partie des 3 millions de victimes annuels de la tuberculose.
b. Autres applications sanitaires et médicales
Il en existe de nombreuses, dans des domaines très variés, mais nous n'en citerons que trois ici.
De la même façon que le test de la tuberculose, la bioluminescence du ver luisant peut être utilisée pour tester la résistance de tumeurs à des anticancéreux, par exemple.
Le gène luc est également utilisé aux Etats-Unis et au Canada pour tester l’innocuité de la viande face à des bactéries comme les salmonelles (Salmonella sp.) ou Escherichia coli. Auparavant, pour savoir si la viande était contaminée, il fallait mettre un morceau de viande en milieu de culture et attendre l'apparition d'une hypothétique colonie bactérienne. Cette méthode nécessitait un temps d'attente de trois jours, pendant lesquels la viande était bien souvent déjà vendue dans les boucheries. On utilise maintenant un vecteur viral (un bactériophage) pour transférer le gène luc dans les bactéries qui nous intéressent. Si la viande est contaminée, elle luit faiblement car le gène luc est traduit et permet la synthèse de la luciférase. Par ce système, on peut détecter la plupart des bactéries responsables de graves maladies alimentaires : E. coli, Salmonella mais aussi Staphylococcus aureus (Staphylocoque doré) ou Listeria monocytogenes responsable d'empoisonnements sanguins sévères ou de méningites. Le gène luc est alors inséré dans un type de bactériophage spécifique à chaque bactérie.
On peut aussi citer pour finir une utilisation écologique : la détection d'ions de mercure et d'aluminium, parmi d'autres, par l'intermédiaire de bactéries à qui on a inséré le gène luc au milieu des gènes de résistance à ces ions. Par exemple, si une bactérie résistante au Hg est en présence de Hg, le gène luc sera activé en même temps que le gène de résistance. Après ajout de luciférine, on peut suivre au luminomètre la contamination de l'eau à l'ion Hg. Cette technique est particulièrement utile pour tester les polluants dans des échantillons d'eau lorsque les concentrations sont trop faibles pour pouvoir être détectées par les moyens conventionnels.